Identifizierung von Mikroorganismen mittels MALDI-MS - Features
In nur drei Schritten zum Ergebnis
Probenvorbereitung
Bereiten Sie zunächst eine Mikroorganismenkolonie (oder Kulturflüssigkeit) vor. Eine vorherige Gram-Färbung oder morphologische Beurteilung ist hier nicht erforderlich. Zudem wird keine spezielle Erfahrung oder Hintergrundwissen über Mikroorganismen benötigt. Die Analyse ist mit wenigen Mikrogramm einer Probe möglich, die etwa 105 Bakterien enthält. Da eine einzige Kolonie analysiert werden kann, behindern Kolonien, die von mehreren Bakterienstämmen auf dem Agarmedium stammen, die Analyse nicht.
Schritt 1
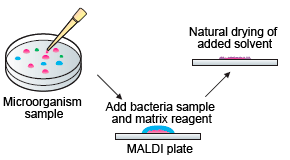
Geben Sie die gesammelte Probe Ihres Mikroorganismus (bspw. Bakterium) und das entsprechende Matrixreagenz auf das MALDI-Target *1. Dabei ist die grundlegende Probenvorbereitung extrem einfach: Mischen Sie einfach die Probe und das Matrixreagenz direkt auf dem MALDI-Target.
Schritt 2
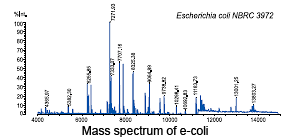
Start der Messung mittels MALDI-TOF MS. Das eindeutige Massenspektrum für die bspw. Bakterienspezies ist schnell ermittelt. Die hier automatische Analyse wird unterstützt mit bereits bestehenden Methoden, wodurch keine Erfahrung in der Massenspektrometrie erforderlich ist. Zudem ist es nicht notwendig, jedes einzelne Massenspektrum zu überprüfen.
Schritt 3
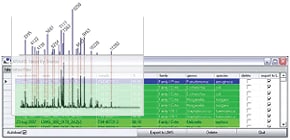
Vergleichen Sie das erfasste Massenspektrum mit den in der Analysesoftware registrierten Spektrendatenbanken, um den Mikroorganismus zu klassifizieren und zu identifizieren. Der gesamte Ablauf von der Datenbanksuche bis zur Anzeige der Identifizierungsergebnisse ist automatisiert. Es sind keine Parametereinstellungen erforderlich.
Zuverlässige Datenbank
Die verlässliche Spektrendatenbank bietet eine sichere Unterstützung zur Identifizierung von Mikroorganismen.
Unübertroffen umfassende Mikroorganismen-Bibliothek
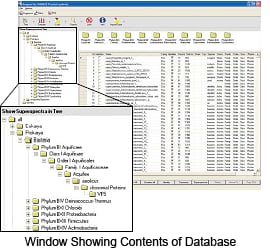
- Vollständig validierte Spektrendatenbank
- SuperSpectraTM Anzahl der registrierten Massenspektren*2: 2.000 min.
Die SuperSpectraTM enthalten Peakgruppen, die als Marker fungieren. Diese Marker-Peakgruppen werden aus mindestens 15 Massenspektren für jeden Mikroorganismus-Stamm extrahiert, die in mehreren Laboren unter vielen Messbedingungen gewonnen wurden. Dadurch wird ein zuverlässiger Nachweis häufig vorkommender Bakterienarten unter verschiedenen Bedingungen ermöglicht.
Ein weiterer Vorteil der SuperSpectraTM liegt in der Reproduzierbarkeit des Massenspektrums für die Identifizierung von Mikroorganismen mittels MALDI-TOF MS. SuperSpectraTM und andere Datenbanken enthalten etwa 40.000 Massenspektren. Die Datenbanken können auch für selten nachgewiesene Bakterienarten angewendet werden.
Individuelle Anpassung der Datenbank für Ihre Bedürfnisse
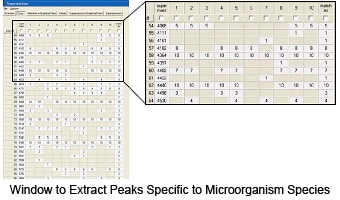
Eine Registrierung eines neuen Mikroorganismus in der Datenbank ist bequem durchzuführen. Die Operationen zur Extraktion von Peakgruppen, die als Marker aus mehreren Massenspektren dienen können, sind einfach und intuitiv. Dies ermöglicht es den Anwender*innen, schnell eine hochwertige Datenbank aufzubauen.
*1 Infektiöse oder möglicherweise infektiöse Proben sind vor der Handhabung stets zu inaktivieren.
*2 Die Gesamtzahl der Massenspektren kann sich aufgrund von Datenbankoptimierungen ändern
Verbesserte Unterscheidungsmöglichkeiten
Strain Solution (option)
Hochpräzise bakterielle Charakterisierung mittels MALDI-Massenspektrometrie
Die Strain Solution-Software verwendet ein MALDI-TOF-Massenspektrometer, das auf der S10-GERMS-Methode basiert, um Bakterien auf Unterarten- oder Stammebene zu unterscheiden.
Die Bakterien können durch die Vorregistrierung von Markern charakterisiert werden, die es dem Benutzer*innen ermöglichen, die Unterarten und Stämme von Bakterien zu unterscheiden. Anschließend werden diese Marker mit den MALDI-TOF MS-Messdaten abgeglichen, um den Zielbakterienstamm zu bestimmen.
Besondere Merkmale
- Präzisere und zuverlässigere Unterscheidungsergebnisse als mit herkömmlichen Methoden zur Identifizierung von Mikroorganismen durch MALDI-TOF-MS-Fingerprinting
- Schnelle Charakterisierung von Bakterien durch genetische Unterschiede in der Basensequenz
- Erstellen und Anpassen von Datenbanken mit Bakterienstämmen
- Durchführung molekularer phylogenetischer Analysen mit externer Software
Anwendungen
Die Software erweitert die Anwendungsmöglichkeiten zur Bakterienidentifizierung, indem sie eine Charakterisierung bis auf Stamm-Ebene ermöglicht. Diese Software ist ideal geeignet für:
- Verwaltung der Sammlung von Bakterienkulturen
- Unterscheidung genetisch ähnlicher Bakterien, die durch 16S rRNA-Gen-Sequenzierung schwer zu unterscheiden sind
- Molekulare phylogenetische Analyse
S10-GERMS*-Methode
Die S10-GERMS-Methode verknüpft Informationen über das Molekulargewicht ribosomaler Proteine, die durch MALDI-TOF MS ermittelt wurden, mit genetischen Informationen zur Klassifizierung und Identifizierung von Mikroorganismen. Die Konzentration auf das S10-spc-alpha Operon, für das etwa die Hälfte der ribosomalen Proteine kodiert ist, ermöglicht die Verbindung von genetischen Informationen mit Molekulargewichtsinformationen, selbst bei nicht genomisch sequenzierten Bakterienarten.
* S10-GERMS: S10-spc-alpha Operon Genkodiertes Ribosomenprotein Massenspektrum
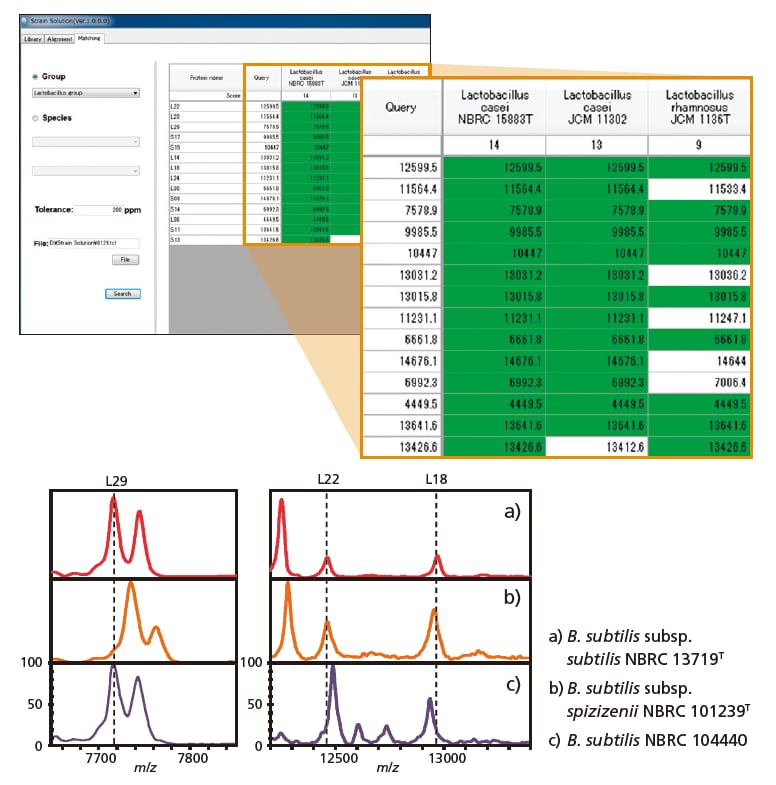
Abbildung: Beispiel für die Unterscheidung von Bakterienstämmen mit 99,9 % 16S rRNA-Gen-Homologie unter Verwendung der S10-GERMS-Methode
Workflow der Strain Solution
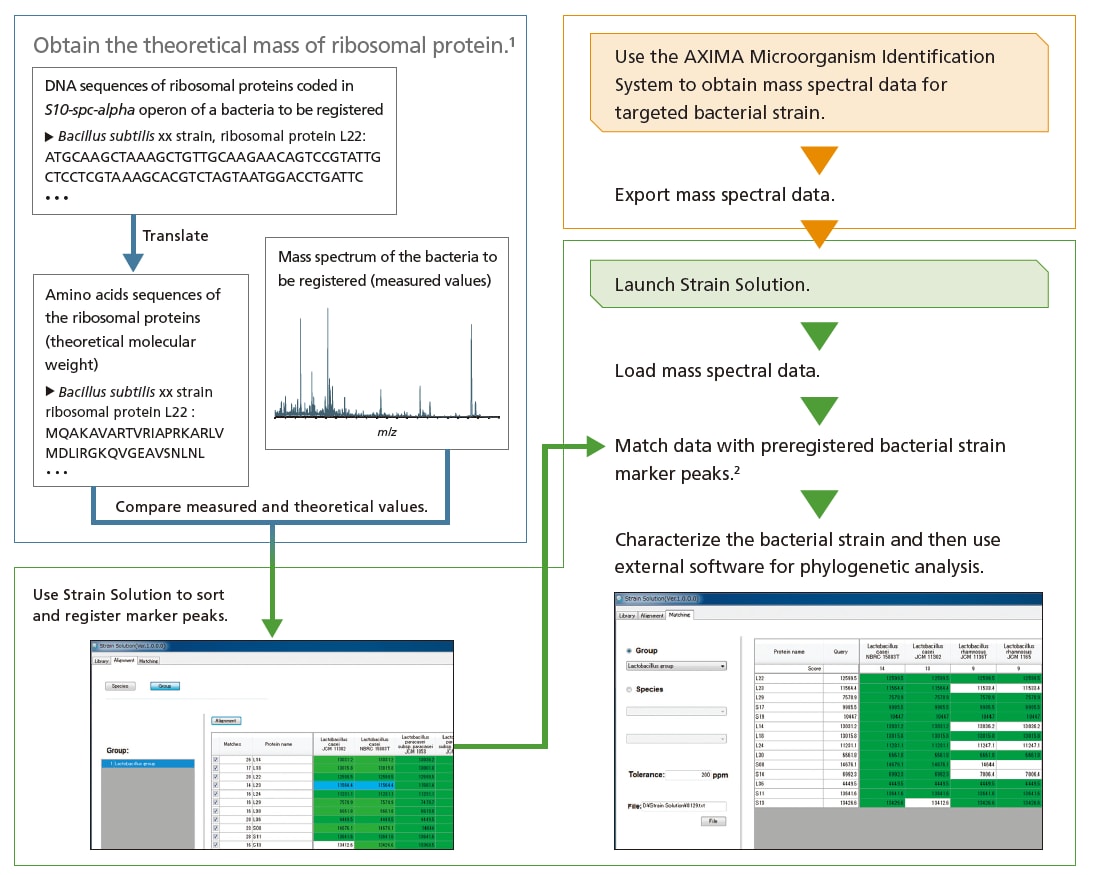
1. Die unter „Obtain the theoretical mass of ribosomal protein“ angegebenen Punkte weisen auf Funktionen hin, die nicht in der Software enthalten sind.
2. Die Software enthält keine Marker-Peaks, die für andere Proben als die Testprobe registriert wurden. Marker-Peaks der Bakterienstämme für die gewünschte Arten müssen im Voraus registriert werden.


