eMSTAT Solution - Features
Statistical Analysis Software for Direct Ionization Mass Spectrometry
Statistischer Analysemodus
Die eMSTAT (easy mass spectrometry statistical) Solution ist eine anwendungsfreundliche statistische Auswertesoftware von MALDI-Daten mit einer Vielzahl an Funktionen sowohl für univariaten und multivariaten Analysen als auch zu einer flexiblen Gruppierung von Probendaten. Vereinfachen Sie ihre statistische Datenauswertung bei Ihren MALDI Analysen.
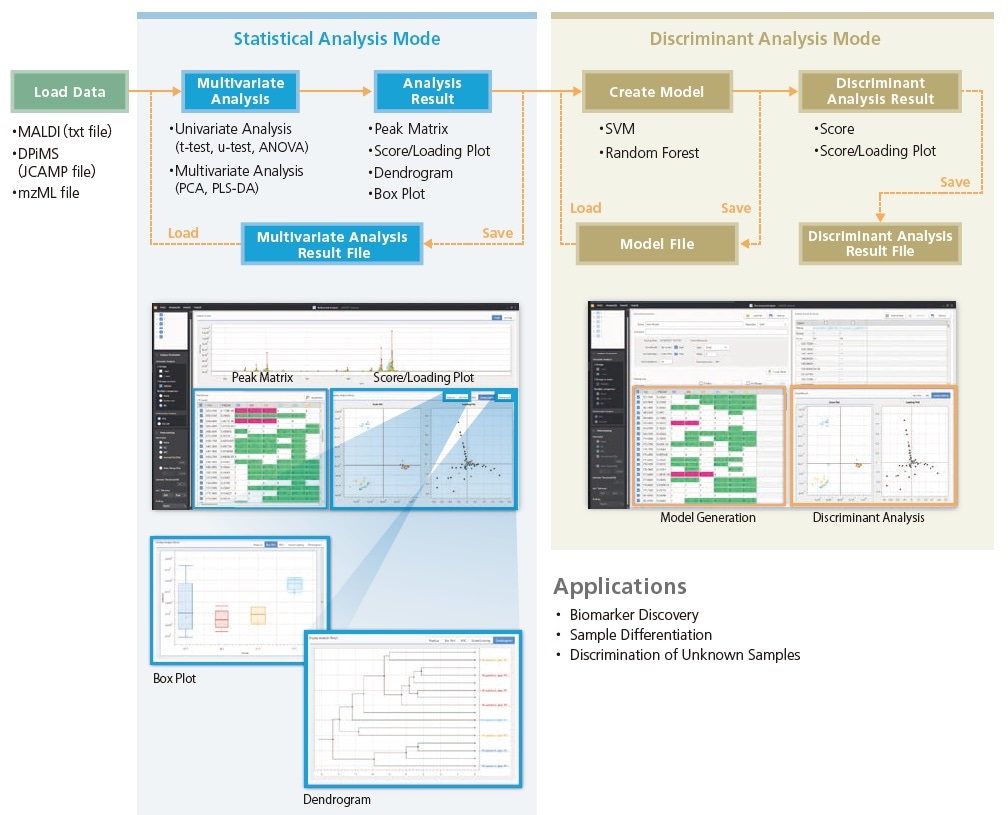
Diskriminanzanalysemodus
Implementieren Sie Ihre Ergebnisse des statistischen Analysemodus, um unbekannte Proben zu unterscheiden.
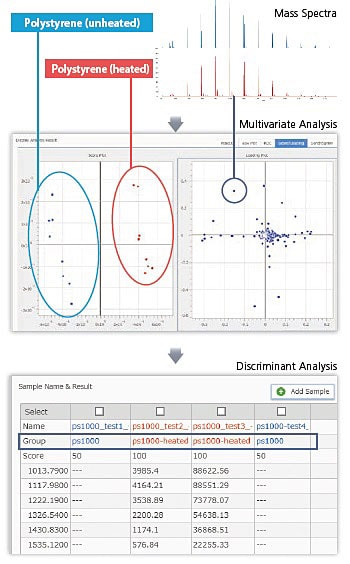
Polystyren-Polymere (erhitzt und nicht erhitzt) wurden mittels einer multivariaten Analyse (PLS-DA) aus dem MALDI-Spektrum in zwei Gruppen (Score Plot), erhitzt und nicht erhitzt, aufgeteilt. Ein Loading Plot erleichtert Ihnen zusätzlich die Bestimmung von Marker Signalen. Die Verwendung solcher identifizierten Marker wird zur Erstellung eines Diskriminanzmodell genutzt und ermöglicht Ihnen durch die übersichtliche Benutzeroberfläche eine korrekte Unterscheidung zwischen allen erhitzten und nicht erhitzten Polymeren.
Durch die Kombination der Software eMSTAT Solution mit der MALDI-Spektrometrie werden Ihre Probenmessungen von großen Molekülen wie Polymere, Proteine, Fette oder Zucker mit einer bequemen statistischen Datenauswertung vereint.

Flexible dynamische Gruppierungsfunktion
Einfache Differenzierung für die Klassifizierung von Rindfleisch
Die flexible Probengruppierung basierend auf registrierten Qualitätsinformationen erleichtert die Entdeckung von Biomarkern.
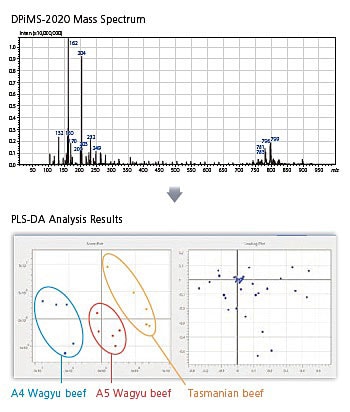
Extrakte aus kommerziellem Rindfleisch (Tasmanisches und Wagyu-Rindfleisch der Güteklasse A5/A4) wurden in einem DPiMS-2020-Massenspektrometer analysiert. Die resultierenden Spektren wurden dann mittels PLS-Diskriminanzanalyse analysiert. Ein Score-Plot bestätigt die Gruppierung in drei Gruppen und ein Loading Plot bestätigt, welche Metabolitenspitzen die Gruppierung beeinflussen.
Mit der eMSTAT-Lösung können Spektren, die durch praktische Metabolitenanalyse in einem DPiMS-2020-Spektrometer gewonnen wurden, verwendet werden, um leicht zwischen Unterschieden in Lebensmitteln, Pflanzen und anderen Proben zu unterscheiden und nach Informationen über Metaboliten zu suchen, die zur Differenzierung beitragen.

Unterstützt eine Vielzahl von Datenformaten aus MALDI-TOF MS
JCAMP-, ASCII- und mzML-Dateneingabe werden unterstützt.


