Multi-omics Analysis Package - Anwendungen
LC/MS, GC/MS Data Analysis Software

Most of the documents on the LITERATURE is available in PDF format. You will need Adobe Acrobat Reader to open and read PDF documents. If you do not already have Acrobat Reader, you can download it free at the Adobe's Website. Click the GET ADOBE READER icon on the left to download a free copy of Adobe Acrobat Reader.
Beispiel für eine Datenanalyse
Verwendung des Lipidmediator-Methodenpakets zur Darstellung quantitativer Veränderungen in menschlichem Plasma und Serum
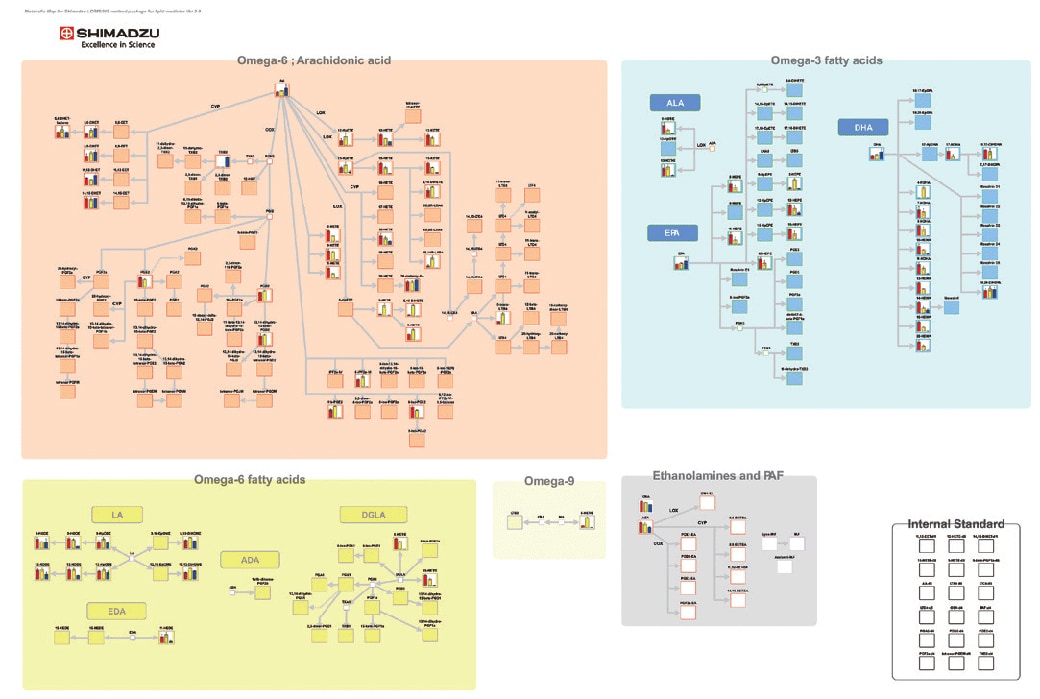
Die obige metabolische karte wurde in Zusammenarbeit mit der Abteilung für Lipidomik der Medizinischen Fakultät der Universität Tokio entwickelt. Weitere Einzelheiten finden Sie unter Application News 01-00250.
Verwendung des Cell Culture Profiling Methodenpaket zur Anzeige von Metaboliten im Zeitverlauf im iPS-Zellkulturüberstand.
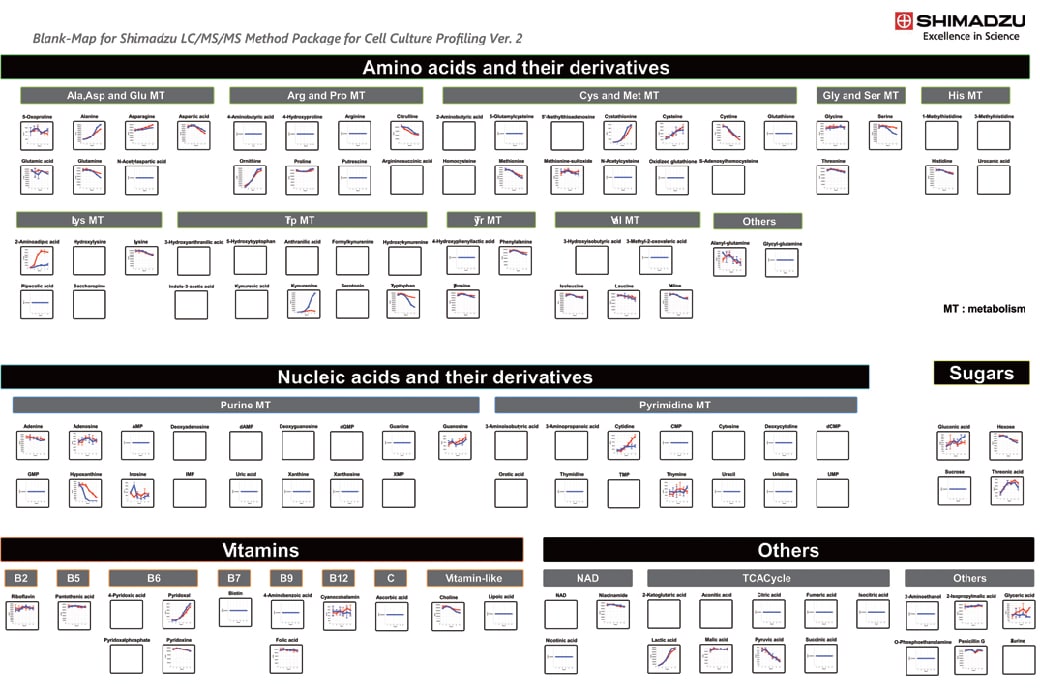
Diese Daten wurden von esearch & Development Center for Cell Therapy, Foundation for Biomedical Research and Innovation, Kobe bereitgestellt. Weitere Einzelheiten finden Sie unter Application News C209.
Umfassende Analyse von Zellextrakten und Kulturüberständen von E coli und Hefe unter Verwendung des Methodenpakets „Primärmetaboliten“.
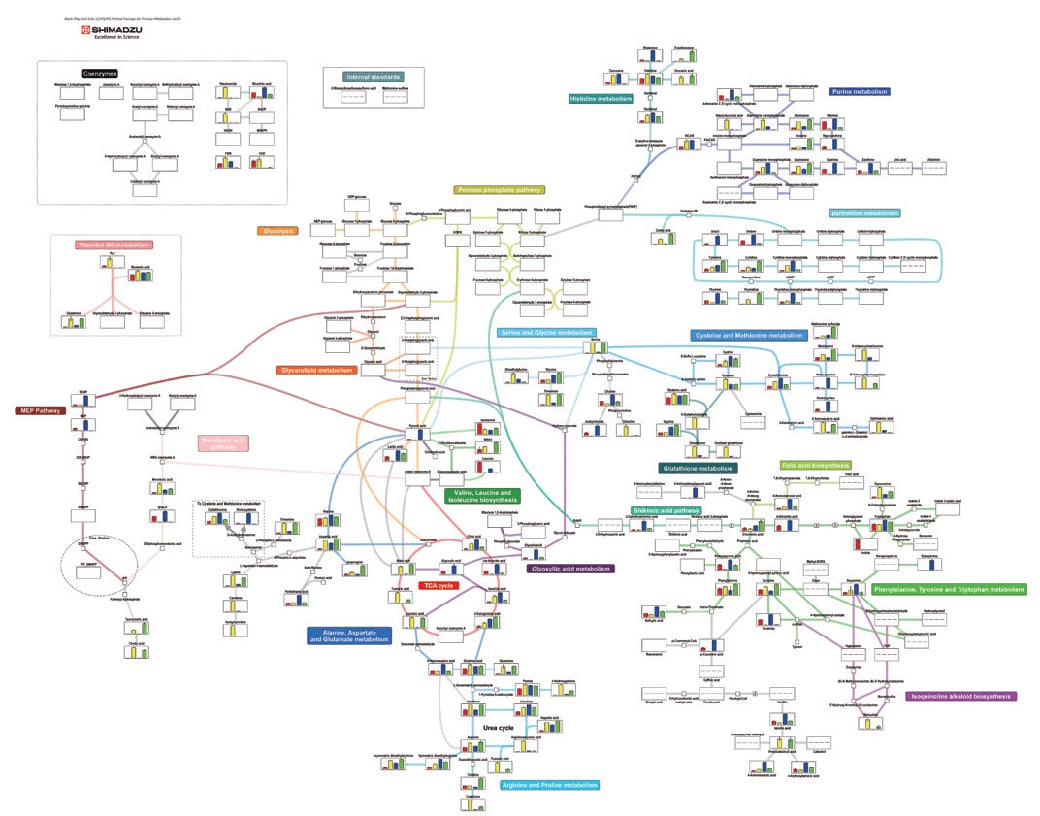
Diese Daten wurden vom Engineering Biology Research Center der Universität Kobe bereitgestellt.
Verwendung des Gallensäure-Methodenpakets zur Analyse von menschlichem Blutplasma und Metaboliten im Mäusekot
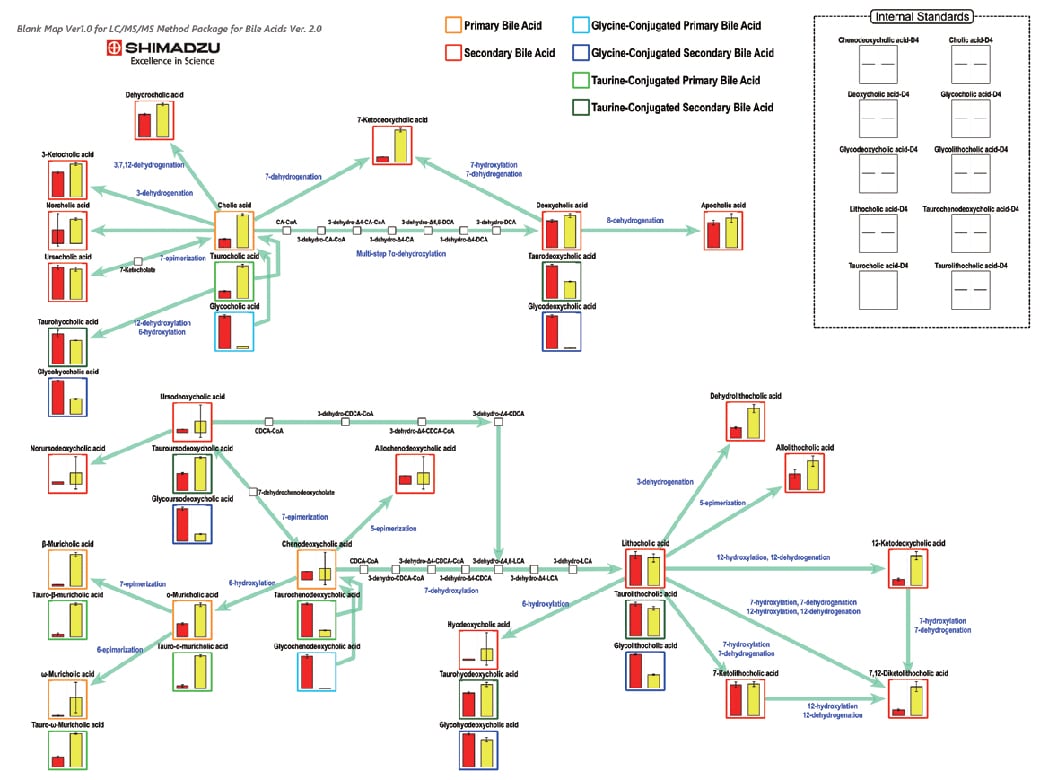
Weitere Einzelheiten finden Sie unter Application News 01-00196.
Analyse metabolomischer Unterschiede in mutierten Drosophila mittels LC-MS und GC-MS – Integrierung von LC-MS- und GC-MS-Daten in metabolische Karten –
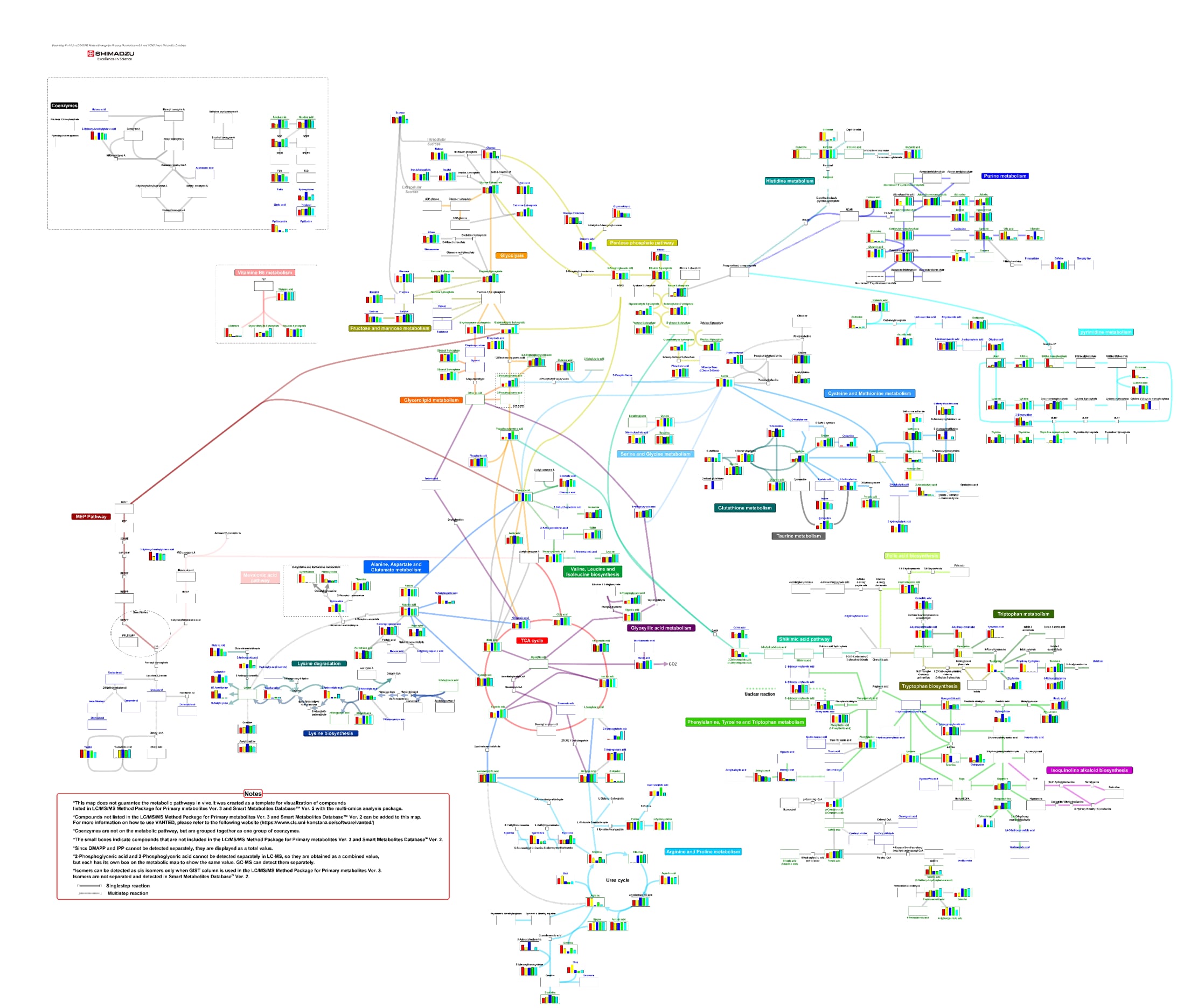
Weitere Einzelheiten finden Sie in den Application News 01-00410 und 01-00491
Tools zur Datenanalyse
Tools zur Datenanalyse
Multi-omics Analysis Package basiert auf Softwaretools (Gadgets genannt), die auf der GARUDA-Plattform veröffentlicht wurden – einer offenen Forschungsplattform, die von der GARUDA Alliance entwickelt und vom Systems Biology Institute, Japan (SBI) geleitet wird.
Zusätzliche Optionen für Ihre Datenanalyse innerhalb des Multi-omics Analysis Package

Volcano Plot
Die Funktion kombiniert einen T-Test und eine Fold Analyse, um Unterschiede zweier Gruppen darzustellen.

EasyStats
Dieses Tool dient zur Visualisierung der Ergebnisse durch die Hauptkomponentenanalyse (PCA), der hierarchischen Clusteranalyse (HCA) oder Boxplots (einschließlich T-Test). Die Ergebnisse der Metaboliten-Datenanalyse können in einem einzigen Fenster angezeigt werden, um eine umfassende Bestimmung charakteristischer Veränderungen zu ermöglichen.

VANTED
Diese Option wurde von der Universität Konstanz betreut. Sie dient zur Visualisierung und zur Analyse von Interaktionen verschiedener Datensätze. (GARUDA-Unterstützung wurde an der Monash-Universität, Melbourne, entwickelt)

Cytoscape
Diese bioinformatische Funktion wurde von dem Cytoscape-Konsortium entwickelt. Sie findet unter anderem zur Visualisierung von Stoffwechselwegen und zur Integration von Genexpressionsprofilen Verwendung. Insbesondere hilft diese Option zur Darstellung von Korrelationen und der Untersuchung von Interaktionen.
Methodenpakete für Ihre Omics-Anwendungen

* Das Multi-Omics-Analysepaket ist im LC-MS/MS-Methodenpaket für Primärmetaboliten, Zellkultur Profiling und Metaboliten enthalten.
* In der Exact Mass Database for Endogenous Metabolites gibt es keine leere Karte zur Visualisierung kurzkettiger Fettsäuren.